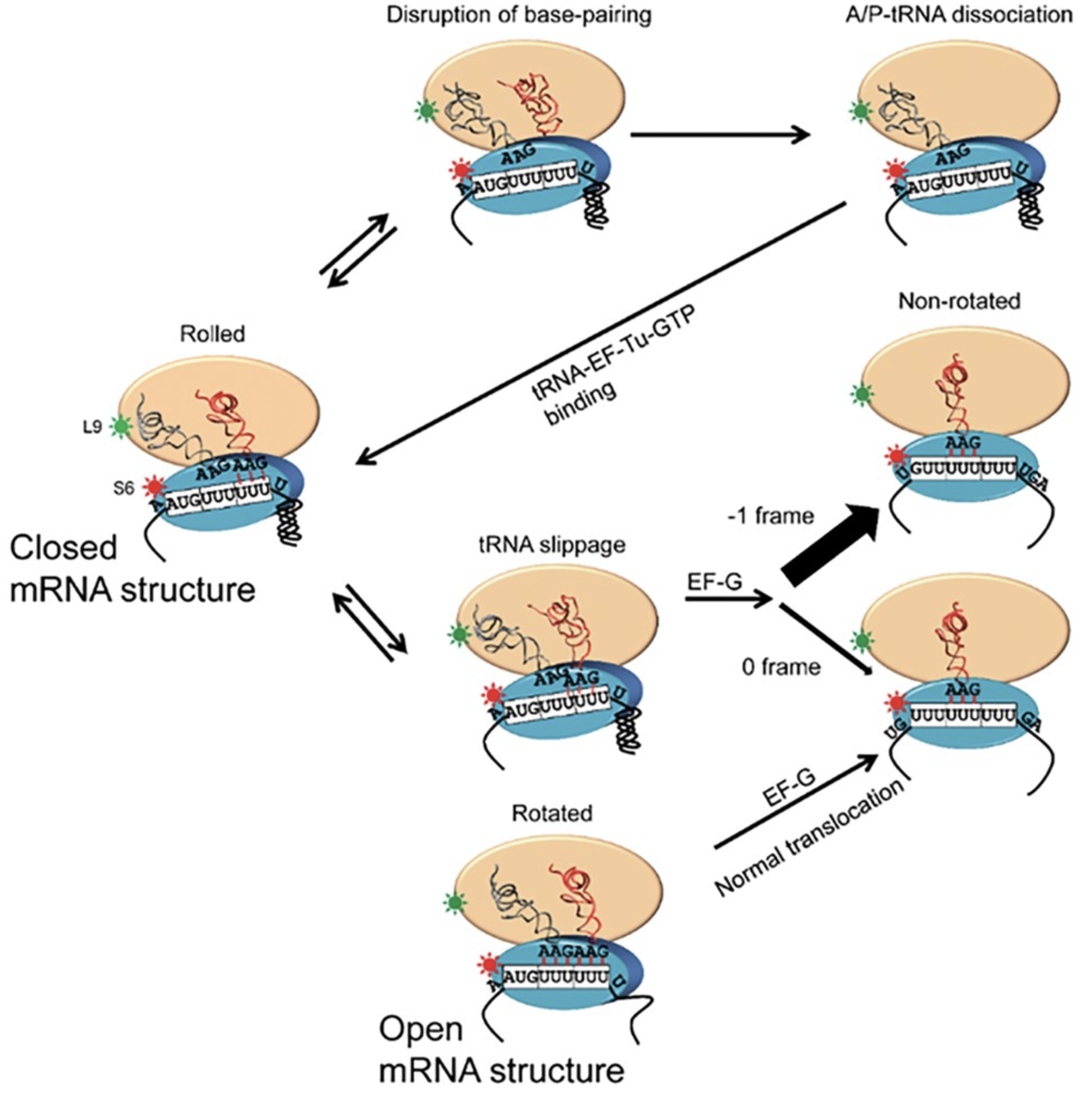
溫進德老師與清大楊立威老師合作團隊發表論文 Resolution-exchanged structural modeling and simulations jointly unravel that subunit rolling underlies the mechanism of programmed ribosomal frameshifting. Bioinformatics. (2019) 35(6): 945 – 952. 我們結合多種分子動態模擬技術,發現 mRNA pseudoknot 的 helical stem 與相鄰 ssRNA loops 之交互作用可形成穩定的摺疊中間構型,並引起核醣體 30S 小次單元的滾動現象,以促進 tRNA 之滑動而導致 programmed ribosomal frameshifting (FS) 。當 loop 序列突變後,儘管 base pairing 仍完整,但其交互作用則無法驅使 tRNA 滑動。模擬結果不僅與突變和單分子實驗結果相符,更完整解釋了 mRNA 結構如何引起核醣體結構變化以及 FS 。